摘要:野生菌种类资源丰富,特别在云南山区农村等地区分布广泛,但因为野生菌种类繁杂,人们缺乏对野生菌进行有效识别分类的手段,每年在云南都会出现许多野生菌误食事件。本设计基于分割后的野生菌图像,对其提取颜色特征、纹理特征,对分类器训练,建设交互式的野生菌图像识别系统。该系统通过交互式野生菌图像的在线识别,可在一定程度上有效避免野生菌误食事件的发生。
关键词:野生菌图像;交互系统;识别
1 引言1.1 选题背景
野生菌即野生的蘑菇,又称大型真菌。野生菌中包括两大种类,一种是人体食用野生菌后身体会产生一系列的毒理反应这种称为毒蕈又叫毒蘑菇,还有一种是无毒的野生菌。我身在云南,这里的野生菌资源极为丰富,从小便是吃着野生菌长大的。野生菌虽然味道鲜美,营养丰富,但是每当野生菌疯狂生长之际野生菌中毒事件便屡见不鲜。针对一些有毒野生菌和无毒野生菌外观相似的情况,目前还没有一种快速,准确和有效的办法能对有毒野生菌进行识别,即便相关专家有时也要通过显微镜观察才能准确地进行种类识别,所以非常容易发生误食野生菌中毒事件[1]。
一个有效且准确的野生菌图像识别系统应用后一定程度上可以很好地提高人们对野生菌种类的准确认识,从而减少人们因为误食有毒野生菌而危害身体健康乃至生命的病例。最近几年,随着抖音等短视频平台的普及,野生菌的美味被越来越多的人所追捧,但是认真科普野生菌种类的视频并不多,一个是野生菌种类繁多不利于人们记忆,二是大部分群众普遍相信自己周围人的经验,看别人吃了几天没事就会相信这野生菌没毒,殊不知有毒野生菌种类中存在会引起慢性肾损害的野生菌,段时间吃没事,然而长时间吃则会导致更严重的肾损害,也因此增加了人们预防迟到有毒野生菌的难度。
本设计通过选取了云南常见的野生菌图像,包括了六大种类,七百多组野生菌图像来搭建系统的数据集。其中,有毒和无毒野生菌各占一半。在误食有毒野生菌导致中毒死亡的事件中,高达90%是因为误食剧毒鹅膏所引起的[2]。考虑后我将本系统的识别种类设置为:变绿红菇、毒蝇伞、黄毒蝇鹅膏菌、红汁小菇、双孢鹅膏菌、血色牛肝菌。我选择原生态环境作为野生菌图像的背景,主要通过各种真菌研究网站采集云南实地的这六种野生菌图像,使其对云南常见野生菌的识别更具说服力。通过自写的算法分割野生菌图像、分割后的野生菌图像提取颜色和纹理特征、SVM支持向量机多分类模型的训练预测以及优化、以及交互式界面的设计、系统测试等建立了一个鲁棒性良好的交互式野生菌图像识别系统实现对这几种云南常见野生菌图像的识别分类。
1.2 国内外研究现状
近几年来,随着互联网技术和机器学习的蓬勃发展。人们在长期采集野生蘑菇的过程中积累了很多对有毒野生菌的识别经验和方法[3]。国内外都出现了许多针对不同数据集,不同地区常见物种所建立的大型真菌识别分类系统。
刘斌等人提出一种基于贝叶斯分类的有毒大型真菌识别方法,选取了八千多组野生菌的23维特征作为数据集达到了98%以上[4]。然而,实际情况下很难直接得到待识别的野生菌23维度特征,往往需要通过一些专业的生理切片观察以及一些其他手段才能提取到野生菌的23维度特征方法略微复杂。
罗宏波等人提出一种有毒野生菌图像的分割方法,是基于水平集复杂背景下的,结合了数字抠图与水平集结合,最后实现对复杂背景下的野生菌图像自动抠图[5]。
庞凤丽开发的基于Android移动端的内蒙古本地有毒野生菌辅助识别系统,提取蘑菇的颜色和纹理作为特征向量,基于BP神经网络对野生菌毒性进行识别[6]。然而,在野外情况下难以准确获得野生菌的特征属性,而且,背景复杂的情况下,识别系统的适应能力和泛化能力会大大降低。
肖杰文等人利用轻量级ShuffleNetV2模型实现自然环境下七种可食用蘑菇图像的分类问题[7]。然而,在实际生活中,其识别的种类鸡枞,灵芝等可食用菌的识别在预防误食有毒野生菌的情况下较难发挥作用,且缺乏一定的交互界面不利于给普通识别蘑菇的使用人员。
近年来,由百度出品的百度识图以及中科院出品的花伴侣都在多端实现了高精度、高效度的植物识别分类,但是其在大型真菌图像的识别上缺乏精度。
综上所述,开发一款基于云南常见野生菌图像的交互式识别系统,其数据集包括了有毒以及无毒的野生菌图像对于预防云南地区的误食有毒野生菌事件发生具有实际性的意义。并且系统数据集种类的选取基于云南常见的野生菌,以及交互界面的设计可以一定程度上提示系统使用人员的识别效率,且在识别之后还能再交互界面给出详细识别信息以及易混淆野生菌种类的信息与图片。
2 图像集的选择与收集
2.1 野生菌种类的选择
本系统旨在识别云南地区常见有毒与无毒的野生菌图像,故选取了共六种有毒和无毒种类的野生菌,考虑到鹅膏菌的高中毒率,我将系统待识别的种类设置为了:变绿红菇、毒蝇伞、黄毒蝇鹅膏菌、红汁小菇、双孢鹅膏菌、血色牛肝菌。
2.2 野生菌图像的采集
本系统的野生菌图像选取了其在复杂生态背景下的野生菌图像进行识别,以达到识别不同环境背景下的野生菌图像。野生菌图像数据的来源则是主要通过各种真菌以及生物研究网站上采集云南实地观察到的这几种野生菌图像,使其对云南本地的野生菌识别更有说服力。
3 野生菌图像预处理
3.1 野生菌图像剪裁
由于野生菌生长环境多变,其背景可能是树林,草地等。为了提高本系统的识别效率,减低野生菌背景图像对本系统的影响,本设计将原始的野生菌图像剪裁为256×256像素的jpeg格式图片。通过将图1多余的一些野生菌图像背景剪裁掉如图2为本系统后续的野生菌图像分割、野生菌图像特征提取做好准备。
图1 剪切前的血色牛肝菌 图2 剪切后的血色牛肝菌
Fig.1 Before Shearing Fig.2 After Shearing
3.2 野生菌图像分割
通过对野生菌图像进行野生菌图像和背景图像分割可以很大程度上保留野生菌占图像的主体性,减少复杂背景的干扰。常见的图像分割方法包括区域分割法、阈值分割法等,我尝试过后发现并不适合本野生菌图像集;以深度学习为代表的一些语义分割算法如U-Net虽然可以实现复杂背景图像下的精准分割,但却离不开大量数据的学习,考虑到本系统的数据量不大并且需要考虑最后交互界面的响应速度,于是我结合邻域运算创建了一个适用于本系统的野生菌图像分割算法。
3.2.1 创建的分割算法
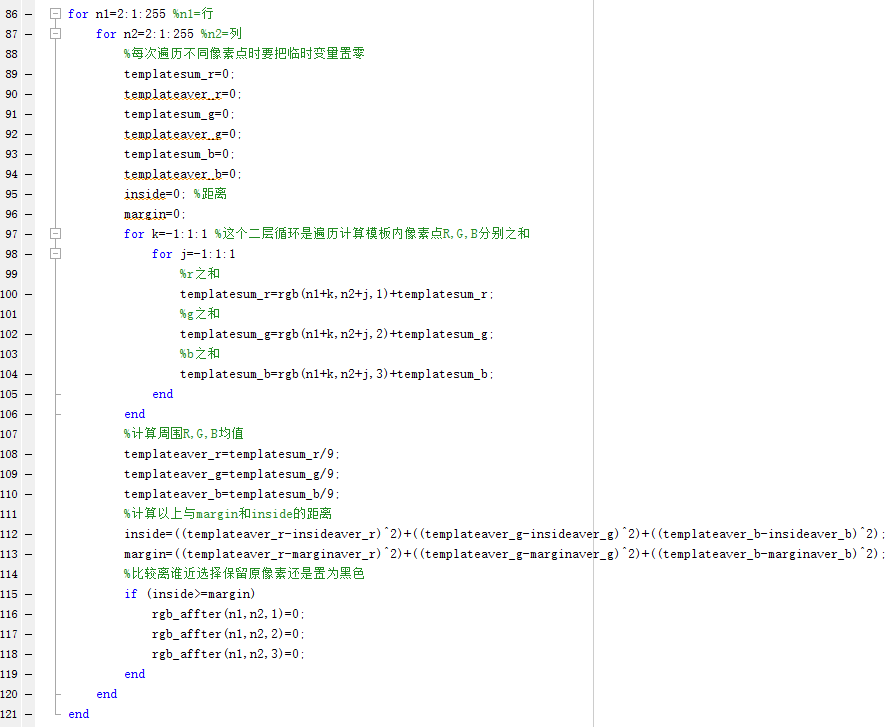
简单概述来说,这个算法原理是:提取野生菌部分像素计算其RGB颜色均值,再提取背景部分像素计算R,G,B均值;随后用3x3模板遍历图像所有像素计算模板颜色均值作为此像素点颜色均值,接着比较此像素颜色空间距离野生菌图像更近还是距离背景颜色空间的距离更近,以此判断此像素为野生菌图像还是背景图像。对的分割之前需要先对图像提取两个评估值:背景三维评估值和核心图像三维评估值。通过计算每个像素点的三维评估值与背景三维评估值与核心图像三维评估值的距离,进而判断此像素点是背景还是野生菌图像的像素。通过在MATLAB测试后我发现HSI颜色空间和RGB颜色空间之间的转换对图像分割影响并不大,于是我采用基于RGB颜色空间进行野生菌图像分割。
(1)背景三维评估值与核心图像三维评估值
基于切割之后野生菌图像的背景是由边缘扩散到野生菌图像周围的特点,我将背景三维评估值的计算范围确定为图像最外围的一圈宽度为三个像素的区域,核心图像三维评估值的计算范围确定为图像中心3x3的区域。评估值的RGB三个分量具体计算公式如式1所示:
`\R=sum_{i=0}^N[R(i)]\/(n+1)`
`\G=sum_{i=0}^N[G(i)]\/(n+1)`
`\B=sum_{i=0}^N[B(i)]\/(n+1)`
(2)基于领域运算的像素点三维评估值
除最外一圈像素点以外,所有像素点的评估值等于以此像素点为中心的3×3均值滤波模板分别对RGB三个分量进行计算。
(3)基于评估值的分割
通过遍历图像计算每个像素的评估值与背景评估值和核心图像评估值的距离,若到背景评估值的距离小于到核心图像评估值的距离,则可以判定此像素点为背景;反之此像素点则为野生菌图像。距离计算公式如式2:
`\D(x,y)=(x_r-y_r)^2+(x_g-y_g)^2+(x_b-y_b)^2`
分割算法的MATLAB核心代码展示如下:
3.2.2 分割算法优化
通过以上步骤后测试对野生菌图像进行分割,然而实际分割效果由于背景复杂等原因,许多的背景并没有被分割掉。在我思考后想到改变图像的对比度和色调等应该可以加强野生菌图像在整个图像中的主体性;于是在野生菌图像分割之前增加其色调和饱和度,通过不断的尝试后我终于找到最佳的色调和饱和度最佳比例,在像素级上实现了较好效果的野生菌图像分割。
图3 分割前的野生菌 图4 直接分割的野生菌
Fig.3 Before Cutting Fig.4 Direct Cutting
图5 优化分割后的野生菌
Fig.5 Optimized Cutting
4 特征工程
野生菌图像的特征提取对对野生菌图像的分类识别有着极其密切的关系,特征提取的方向很大程度上会影响分类器的是被效率。一组好的特征可以直接提高野生菌图像的分类准确率。在观察了大量不同种类的生态野生菌图像后,我发现不同野生菌的形状对其分类结果影响不大,反而是颜色特征和纹理特征会对野生菌的分类产生较大的影响,于是我选取了野生菌图像的颜色特征和纹理特征作为提取值。
4.1 颜色特征
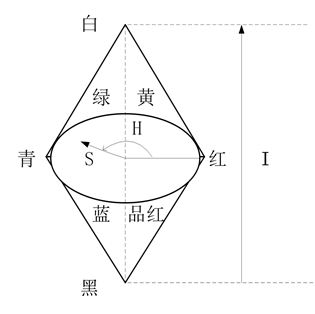
图6 HSI颜色空间模型
Fig.6 HSI Color Space Model
不同的颜色模型对提取后颜色特征建立的分类器效果有着不同的影响,一般来说HSV和HSI颜色模型更加适合模式识别图像分类。所以我选择了基于HSI颜色模型所提取的颜色特征。选择HSI颜色模型进行颜色特征参数的提取,提取其色度、饱和度、亮度三个特征[8]。HSI三个分量则可以通过RGB的值分别计算得到。
分别对分割后野生菌图像的H、S、I颜色通道计算均值、方差、偏斜度一共9个维度作为提取后的颜色特征向量。计算公式分别如下:
`\mu_i=sum_{j=1}^n[P_(ij)]\/n`
`\sigma_i=(sum_{j=1}^n[(P_(ij)-mu_i)^2]\/n)^(1/2)`
`\S_i=(sum_{j=1}^n[(P_(ij)-mu_i)^3]\/n)^(1/3)`
其中Pij为第I个HSI颜色通道下的第J个像素点的值。在此系统的数据集中一共有726组数据其中三种有毒野生菌和三种无毒野生菌,部分有毒和无毒野生菌图像的元素特征展示如图7:

图7 部分无毒野生菌图像提取到的颜色特征
Fig.7 Color Characteristics of non-Toxic Mushrooms

图8 部分有毒野生菌图像提取到的颜色特征
Fig.8 Color Characteristics of Toxic Mushrooms
4.2 纹理特征
4.2.1 灰度共生矩阵
在不同的纹理特征中,灰度共生矩阵属于比较常用和运用比较多的方法。对于取好的距离d和方向θ,在方面为θ的直线上一个像素的灰度值为i,另外一个与它距离为的d的像素灰度值为j的点出现的频数则作为这个矩阵的第ij元素的值。d和θ的取值会产生不同的GLCM,一般d只取少数的值,θ则是取`\0^2`、`\45^2`、`\90^2`、`\135^2`。
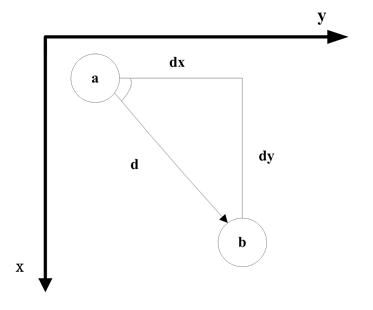
图9 GLCM的像素关系
Fig.9 Pixel Relationship of GLCM
4.2.2 灰度共生矩阵的二次统计量
由灰度共生矩阵则可以产生二次统计量。一般情况都是采用GLCM的二次统计量来作为纹理特征,以达到区分不同图像的目的。GLCM的二次统计量也是非常的多,在灰度共生矩阵的多个特征参数中,有4个特征是不相关的,这4个特征不仅便于计算,且分类精度较高[9]。这四个特征分别为:能量、惯性矩、熵、相关。设在给定的θ和d参数下的共生矩阵的元素已归一化成为频率,并记为P(i,j)。对应的四二次统计量计算公式如下:
(1)能量。灰度共生矩阵的能量即是每个矩阵元素的平方和,当纹理粗时能量较大。
`\N1=sum_{i}sum_{j}P(i,j)^2`
(2)惯性矩。当纹理较粗时惯性矩N2较小,当纹理较细时惯性矩N2较大。
`\N2=sum_{i}sum_{j}(i-j)^2P(i,j)`
(3)熵。当纹理较粗时熵N3较小,当纹理较细时熵N3较大。
`\N3=-sum_{i}sum_{j}P(i,j)logP(i,j)`
(4)相关。如果图像中区域之间灰度值之间相差很大时,相关值会越小。如果图像区域灰度值均匀相等时,相关值相对较大。
`\N4=sum_{i}sum_{j}((i*j)P(i,j)-\overline x*\overline y)/(\sigma_i\sigma_j)`
其中
`\overline x=sum_{i}isum_{j}P(i,j)`
`\overline y=sum_{j}jsum_{i}P(i,j)`
`\sigma_i^2=sum_{i}(i-\overline x)^2sum_{j}P(i,j)`
`\sigma_j^2=sum_{j}(j-\overline y)^2sum_{i}P(i,j)`
本系统提取的纹理特征则是基于这四个二次统计量,分别对每一幅分割后的野生菌图像计算其四个GLCM二次统计量的均值和方差一共八维度的特征向量。图十和图十一是对726幅野生菌图像通过MATLAB实现提取纹理特征后的特征向量。
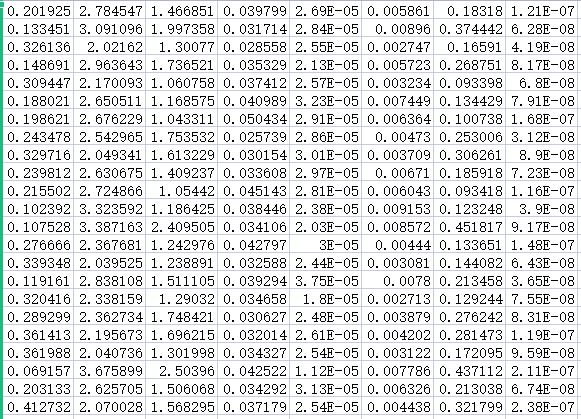
图10 部分无毒野生菌图像提取到的纹理特征
Fig.10 Texture Characteristics of some non-Toxic Mushrooms
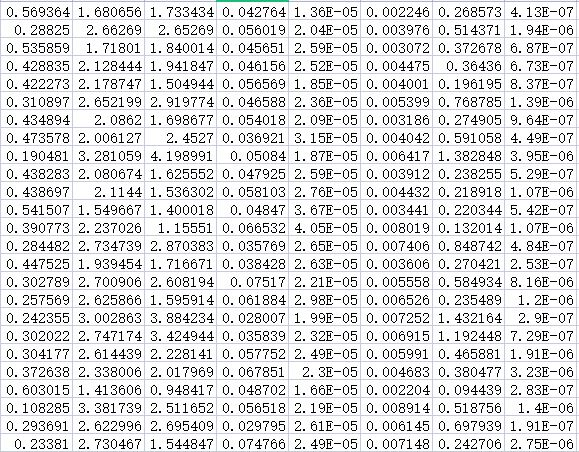
图11 部分有毒野生菌图像提取到的纹理特征
Fig.11 Texture Characteristics of some Toxic Mushrooms
5 支持向量机SVM模型
5.1 SVM概述
支持向量机SVM是由俄罗斯著名统计学家和数学家弗拉基米尔·万普尼克提出的,并且还在此基础上提出了严密的统计学习理论。原理是在所有能够正确实现分类的权向量w里面取求到分类间隔最大的的,就是对线性分类器优化求解的一个很好的指标。Vapnik通过引入结果风险最小化及VC维和松弛变量和核函数,使SVM在面对线性不可分问题时也能取得很好的分类效果和泛化能力。分类间隔d是由距离决策边界最近的那些少量样本决定的,这些样本便称为支持向量Support Vector。
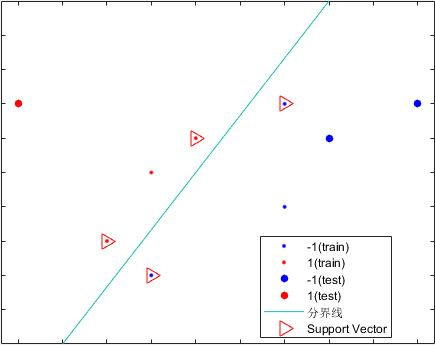
图12 SVM原理展示
Fig.12 Principle Display of SVM
5.2 支持向量机SVM模型的建立
支持向量机SVM模型的建立主要包括训练与测试的数据集和标签集、分类模式、损失函数、核函数、核函数的gamma参数的设置等。
5.3 SVM模型的MATLAB实现
通过在MATLAB中引入LIBSVM工具箱可以易于支持向量机模型的建立以及对SVM模型的调试和参数选择。
5.3.1 SVM支持向量机模型参数选择
支持向量机SVM模型中的损失函数参数-c和核函数参数-g会对模型的识别结果影响较大,这里采取交叉验证(Cross Validation)确定参数-c和-g的方法来选择最优的交叉验证参数。其他参数如核函数则采用默认的参数RBF(径向基)核函数。交叉验证(Cross Validation)参数为5时确定c和g参数的代码:

5.3.2 SVM模型计算步骤
SVM在面对线性不可分问题时需要广义线性化将低维空间映射到高维空间,使其线性可分。通过对数据进行归一化不仅可以提高SVM训练效率还可以减少训练时间并且提高分类精度[10]。因此在进行模型训练之前需要对数据进行归一化预处理,这里选择了MATLAB中自带的MAPMINMAX函数将数据归一化到01区间。图十三为SVM模型计算步骤:
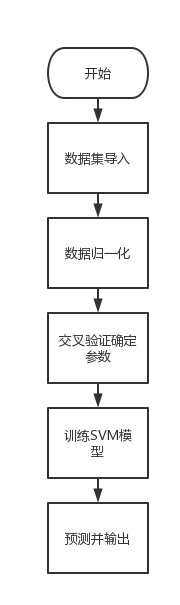
图13 SVM模型计算步骤
Fig.13 Calculation Steps of SVM Model
5.3.3 分类结果的分析
在构建好参数的以及结构的支持向量机SVM模型中,将以上步骤提取到的17维特征向量归一化后输入到模型中,其中训练集共600组有毒和无毒野生菌数据,测试集一共126组有毒和无毒野生菌数据。对训练好的SVM模型输入待测试的126组数据后,识别的结果如图14:
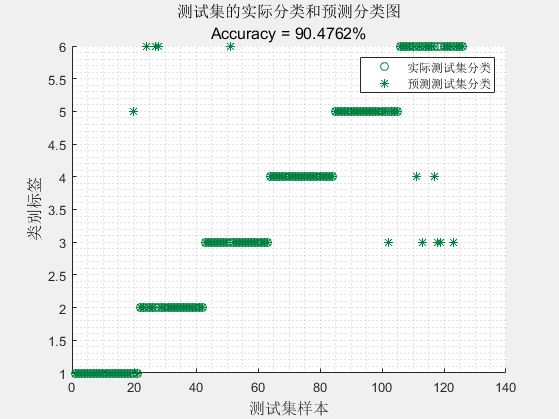
图14 模型预测准确率
Fig.14 Accuracy of Model Prediction
其中,横竖方向分别对应的信息是数据样本数统计结果和识别的种类,对野生菌样本的统计结果如表1所示:
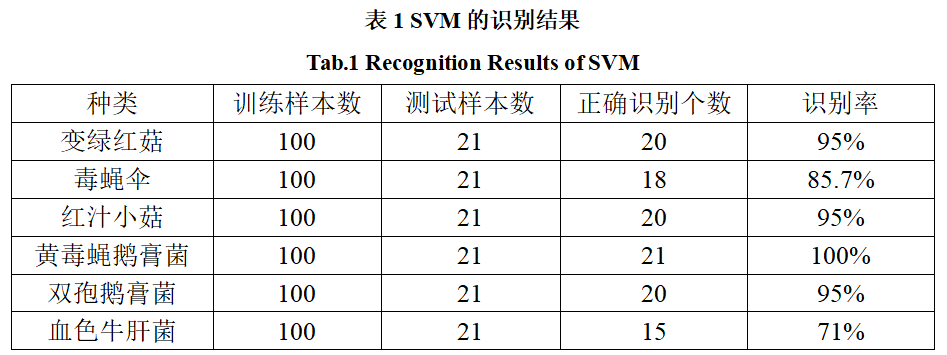
由统计结果可知,在126个有毒和无毒野生菌测试样本中,识别正确了114组野生菌样本,识别率为90.4%,具有较好的识别效果,建立的SVM模型可用于野生菌分类。
6 交互式野生菌图像识别系统详细设计与实现
6.1 主界面
交互界面的设计与完成是基于MATLAB的APPDESIGNER功能模块完成的。交互式野生菌图像识别系统主页面左上侧设有菜单栏,主页面左侧则是待识别的野生菌图像选择和展示区域,主界面右侧是对野生菌图像的识别结果和类似物种信息的展示。通过各种控件完成静态部分的设计,如图15所示。
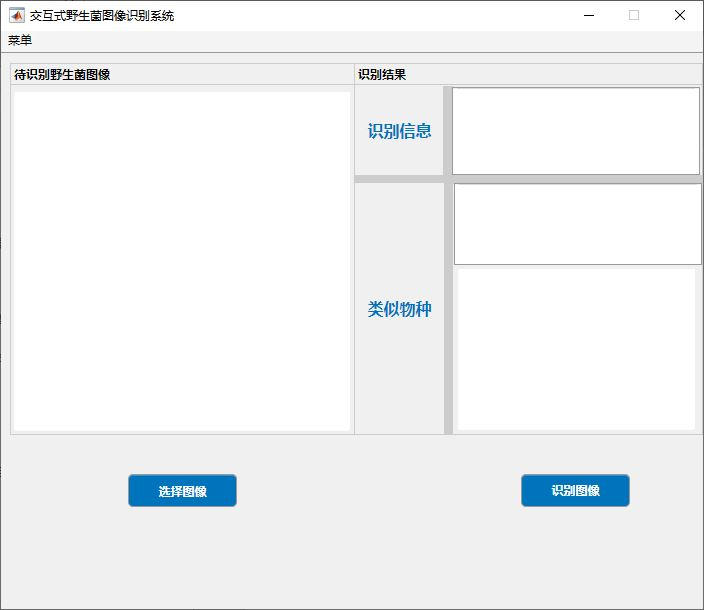
图15 交互主界面
Fig.15 Interactive Main Interface
6.2 交互系统运行识别流程
通过将设计好的的分割野生菌图像函数功能、提取野生菌图像特征功能、由识别种类展示相识种类信息功能模块封装在对应的函数中,有利于在主界面中调用各种功能模块完成野生菌图像系统的识别。识别图像的点击回调函数设计:
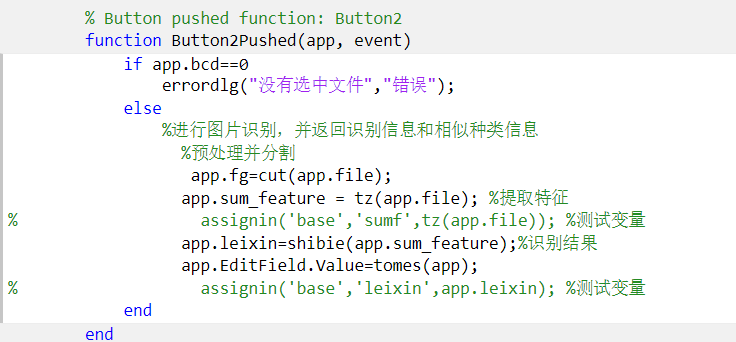
各功能模块之间需要传递的各种参数则是采用全局变量进行传递,在用户选择了需要识别的野生菌后系统的流程如图16所示。
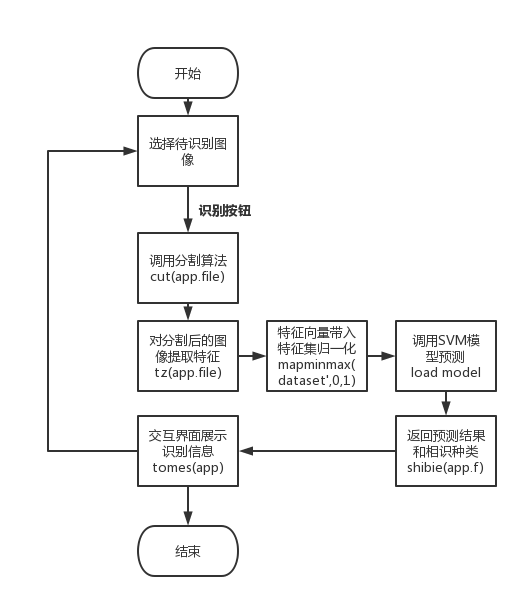
图16 交互系统识别流程
Fig.16 Identification Process of Interactive System
7 交互式系统的测试
在系统的测试中主要采用逻辑驱动测试。在编写程序过程中,各模块功能都出现过错误,通过设置断点,逐步缩小范围,找到问题出现的原因,继而寻求解决方法,最终解决了问题。其中有个BUG是识别结果的不准确,在测试后才发现是逻辑上的错误,由于对待识别野生菌图像提取特征后没有进行归一化就带入了模型识别导致了识别结果的错误,在归一化后就解决了。交互系统开发基本完成后,通过对测试集中的野生菌图像逐一进行识别,实现对系统的整体的测试,测试结果也在此证明系统的识别率在90%以上。
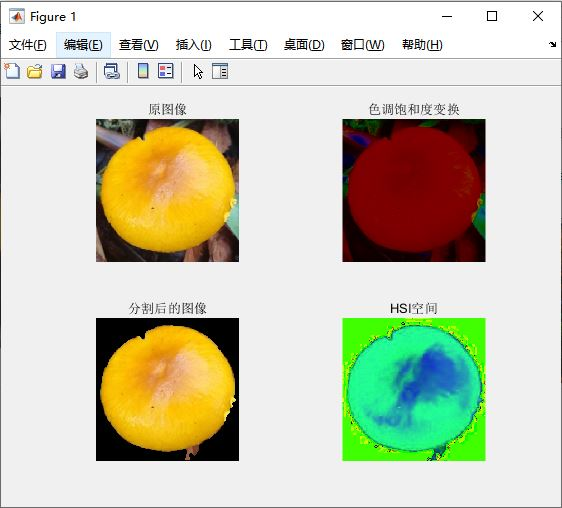
图17 黄毒蝇鹅膏菌识别结果1
Fig.17 Identification Results I
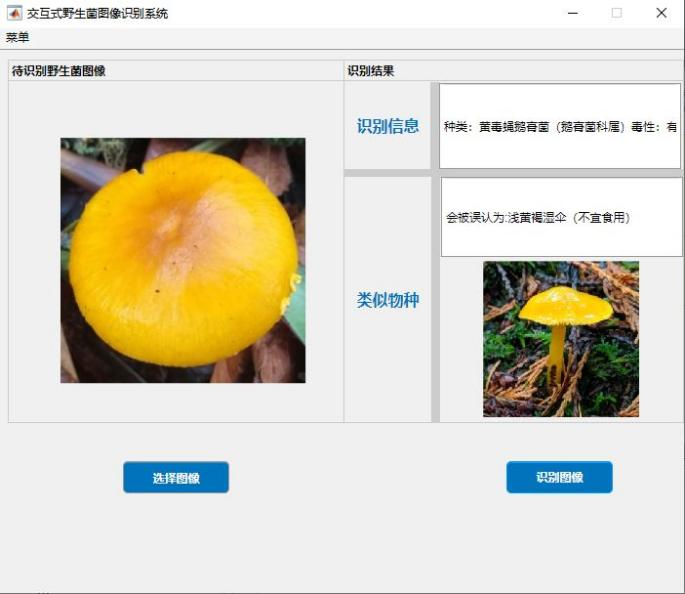
图18 黄毒蝇鹅膏菌识别结果2
Fig.18 Identification Results II
8 结束语
本系统是针对云南地区一些常见野生菌图像开发的具有一定实用性的交互式识别系统,该系统主要实现对野生菌图像识别,旨在为野生菌的研究人员提供一定的帮助也为人们提供一定的警示作用,未知的野生菌最好不要食用。本设计的主要研究内容为:
(1)野生菌生态图像的背景分割,通过各种探索后,我结合数据集构建了一个适用本系统的分割算法,思路是可取的,然而许多细节方面还仍待提高。
(2)野生菌图像的特征提取,结合人眼所观察与判断到的特征,我提取了野生菌的颜色和纹理特征,实验证明颜色和纹理特征的提取对野生菌图像的识别具有较好的效果。
(3)SVM分类模型的建立与优化,通过数据归一化和五折交叉验证确定参数实现了模型分类效果的优化
(4)交互界面的设计,基于MATLAB的APPDESIGNER功能,将系统的各个功能模块整合,交互界面也有利于使用人员的操作。
基于以上的研究,野生菌图像的综合识别准确率达到了90.4%,然而系统的识别率距离完全识别正确还距离很远的路,只有无限接近100的识别率才能让系统真正的走出实验室,走到人们的生活中去。本系统若要继续优化可从以下方面进行研究:
(1)扩大识别的种类和数据。目前系统的数据基于云南常见的野生菌种类,我国上现在已知的野生菌种类是非常多的,通过引入更多的种类和数据以提高系统的实用性。
(2)算法模型的选择。随着系统数据不断的扩大,需要采用一些轻量级的深度学习网络识别模型来提高准确率。
(3)优化分割算法。在生态背景更加复杂的情况下,本系统的分割算法无法提供更加准确的分割效果,可以考虑采用深度学习的语义分割网络来进行分割。
(4)实现手机端和电脑端的轻松访问。本系统目前只支持在Windows下实现对野生菌图像的识别,还不能在手机上识别,这是一个研究方向,可能需要采用其他的平台进行系统设计让系统更加完善。
参考文献
[1] 李林静,李高阳,谢秋涛.毒蘑菇毒素的分类与识别研究进展[J].中国食品卫生杂志,2013,25(4):383-387.
[2] 中国科学院昆明植物所剧毒蘑菇研究取得新进展[J].湖北农业科学,2016,55(21):5729.
[3] J.Ben Schafer,Joseph Konstan,John Riedl.Recommender systerns in ecommerce.ACM C-onference on Electronic Commerce(EC99)[C].USA:Association for Computing Machine-ry,1999,158~166.
[4] 刘斌,张振东,张婷婷.基于贝叶斯分类的毒蘑菇识别[J].软件导刊,2015,14(11):60-64.
[5] 罗宏波,曾海欧,周苏娟,黄展鹏,蒋世忠.基于水平集复杂背景下毒蘑菇图像的提取[J].自动化与信息工程,2015,36(2):27-29.
[6] 庞凤丽.基于Android的毒蘑菇识别系统研究与设计[D].内蒙古:内蒙古工业大学,2019.
[7] 肖杰文,赵铖博,李欣洁,刘钟钰,庞博,杨彝华,王建新.基于深度学习的蘑菇图像分类研究[J].软件工程,2020,23(07):21-26.
[8] 袁向敏. 多标签图像数据的特征选择问题[D]. 温州:温州大学, 2017.
[9] 赵鹏,韦兴竹.基于多特征融合的田间杂草识别分类[J].农业机械学报,2014,(3):275-281.
[10] 汤荣志,段会川,孙海涛.SVM训练数据归一化研究[J].山东师范大学学报(自然科学版),2016,31(04):60-65.
Interactive Wild Mushroom Images Recognition System
Abstract: Wild mushroom is rich in resources, especially widely distributed in mountainous and rural areas in Yunnan. However, because of the large variety of wild mushroom, people lack the means to effectively identify and classify wild mushroom. There are many incidents of wild mushroom ingestion in Yunnan every year. This design is based on the segmented wild mushroom image, extracts it’s color features, SIFT features and PCA dimensionality reduction, trains the classifier, and builds an interactive wild mushroom image recognition system. The system can effectively avoid the occurrence of the wild mushroom misfeeding event through the online identification of the interactive wild mushroom images.
Keywords:Wild mushroom images;Interactive system;Recognition



